[←][→] ath
| functional annotation | |||||||||||||||||||||||||||||||
| Function | F-box/RNI-like superfamily protein |


|
|||||||||||||||||||||||||||||
| GO BP |
|
||||||||||||||||||||||||||||||
| GO CC |
|
||||||||||||||||||||||||||||||
| GO MF |
|
||||||||||||||||||||||||||||||
| KEGG | |||||||||||||||||||||||||||||||
| Protein | NP_001154649.1 NP_001326309.1 NP_001326310.1 NP_001326311.1 NP_001326312.1 | ||||||||||||||||||||||||||||||
| BLAST | NP_001154649.1 NP_001326309.1 NP_001326310.1 NP_001326311.1 NP_001326312.1 | ||||||||||||||||||||||||||||||
| Orthologous | [Ortholog page] AT2G04230 (ath) AT2G26860 (ath) AT3G26920 (ath) AT3G26930 (ath) AT3G49020 (ath) AT3G49030 (ath) AT3G49480 (ath) AT3G50710 (ath) AT3G51530 (ath) AT3G52680 (ath) AT3G52690 (ath) AT3G54910 (ath) AT4G09920 (ath) AT4G10400 (ath) AT4G10410 (ath) AT4G10420 (ath) AT4G13965 (ath) AT4G15060 (ath) AT4G00315 (ath) AT4G00160 (ath) AT4G26340 (ath) AT4G26350 (ath) AT5G38565 (ath) AT5G38570 (ath) AT5G38580 (ath) AT5G38590 (ath) AT5G50270 (ath) AT5G56325 (ath) AT5G56370 (ath) AT5G56380 (ath) AT5G56390 (ath) AT5G56400 (ath) AT5G56410 (ath) AT5G56420 (ath) AT5G56440 (ath) AT5G56560 (ath) AT5G56570 (ath) AT5G56690 (ath) AT5G56700 (ath) AT5G56800 (ath) AT5G56810 (ath) AT5G56820 (ath) AT5G60610 (ath) AT5G62970 (ath) AT1G32375 (ath) AT1G50980 (ath) AT1G51055 (ath) AT1G55030 (ath) AT1G55660 (ath) AT5G56452 (ath) LOC11412326 (mtr) LOC11413726 (mtr) LOC11413807 (mtr) LOC11413983 (mtr) LOC11414794 (mtr) LOC11415711 (mtr) LOC11416055 (mtr) LOC11419778 (mtr) LOC11421874 (mtr) LOC11423645 (mtr) LOC11423850 (mtr) LOC11424583 (mtr) LOC11425125 (mtr) LOC11425566 (mtr) LOC11426644 (mtr) LOC11426664 (mtr) LOC11427307 (mtr) LOC11430133 (mtr) LOC11431527 (mtr) LOC11431961 (mtr) LOC11431962 (mtr) LOC11432072 (mtr) LOC11432126 (mtr) LOC11432164 (mtr) LOC11432207 (mtr) LOC11432426 (mtr) LOC11432817 (mtr) LOC11433505 (mtr) LOC11435452 (mtr) LOC11435524 (mtr) LOC11437196 (mtr) LOC11438287 (mtr) LOC11438558 (mtr) LOC11438844 (mtr) LOC11441487 (mtr) LOC11441934 (mtr) LOC11442468 (mtr) LOC11443901 (mtr) LOC11444775 (mtr) LOC11444992 (mtr) LOC11445048 (mtr) LOC11446820 (mtr) LOC25479526 (mtr) LOC25481141 (mtr) LOC25481193 (mtr) LOC25481575 (mtr) LOC25482939 (mtr) LOC25484041 (mtr) LOC25490043 (mtr) LOC25491096 (mtr) LOC25494331 (mtr) LOC25495280 (mtr) LOC25495678 (mtr) LOC25495688 (mtr) LOC25496353 (mtr) LOC25497957 (mtr) LOC25497960 (mtr) LOC25497965 (mtr) LOC25497980 (mtr) LOC25497981 (mtr) LOC25497982 (mtr) LOC25498231 (mtr) LOC25498241 (mtr) LOC25498279 (mtr) LOC25498281 (mtr) LOC25498312 (mtr) LOC25499685 (mtr) AT3G49040 (ath) LOC100780535 (gma) LOC100792817 (gma) LOC100794386 (gma) LOC101264417 (sly) LOC102661462 (gma) LOC102661539 (gma) LOC102665056 (gma) LOC102667671 (gma) LOC102669752 (gma) LOC103832496 (bra) LOC103833442 (bra) LOC103837190 (bra) LOC103840156 (bra) LOC103841196 (bra) LOC103841395 (bra) LOC103841399 (bra) LOC103841400 (bra) LOC103841402 (bra) LOC103841456 (bra) LOC103841459 (bra) LOC103841937 (bra) LOC103843504 (bra) LOC103844208 (bra) LOC103845079 (bra) LOC103845090 (bra) LOC103846133 (bra) LOC103847623 (bra) LOC103850768 (bra) LOC103851807 (bra) LOC103851809 (bra) LOC103851816 (bra) LOC103851827 (bra) LOC103851828 (bra) LOC103851829 (bra) LOC103851831 (bra) LOC103851832 (bra) LOC103851835 (bra) LOC103851921 (bra) LOC103851922 (bra) LOC103851923 (bra) LOC103852111 (bra) LOC103852136 (bra) LOC103852138 (bra) LOC103852139 (bra) LOC103856768 (bra) LOC103856796 (bra) LOC103856797 (bra) LOC103856799 (bra) LOC103856811 (bra) LOC103856915 (bra) LOC103856923 (bra) LOC103856925 (bra) LOC103856926 (bra) LOC103856929 (bra) LOC103856930 (bra) LOC103856936 (bra) LOC103857150 (bra) LOC103857151 (bra) LOC103857152 (bra) LOC103857209 (bra) LOC103861622 (bra) LOC103864542 (bra) LOC103864611 (bra) LOC103864657 (bra) LOC103864754 (bra) LOC103865921 (bra) LOC103866077 (bra) LOC103866090 (bra) LOC103866312 (bra) LOC103866315 (bra) LOC103868551 (bra) LOC103871266 (bra) LOC103871272 (bra) LOC103871677 (bra) LOC103871678 (bra) LOC103873678 (bra) LOC103875264 (bra) LOC106795655 (gma) LOC106797202 (gma) LOC107275344 (osa) LOC108868815 (bra) LOC108870128 (bra) LOC108870613 (bra) LOC109118850 (sly) LOC112416099 (mtr) LOC112416383 (mtr) LOC112416398 (mtr) LOC112416553 (mtr) LOC112416630 (mtr) LOC112420448 (mtr) LOC112420449 (mtr) LOC112421930 (mtr) | ||||||||||||||||||||||||||||||
| Subcellular localization wolf |
|
||||||||||||||||||||||||||||||
| Subcellular localization TargetP |
|
||||||||||||||||||||||||||||||
| Gene coexpression | |||||||||||||||||||||||||||||||
| Network*for coexpressed genes |
|
||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Coexpressed gene list |
[Coexpressed gene list for AT3G26922] | ||||||||||||||||||||||||||||||
| Gene expression | |||||||||||||||||||||||||||||||
| All samples | [Expression pattern for all samples] | ||||||||||||||||||||||||||||||
| AtGenExpress* (Development) |
257013_at
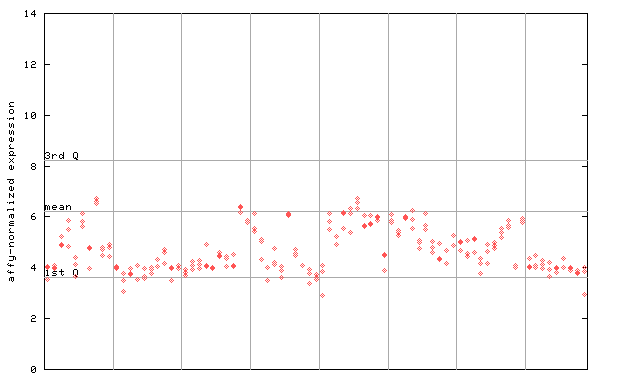
X axis is samples (pdf file), and Y axis is log2-expression. |
||||||||||||||||||||||||||||||
| AtGenExpress* (Stress) |
257013_at
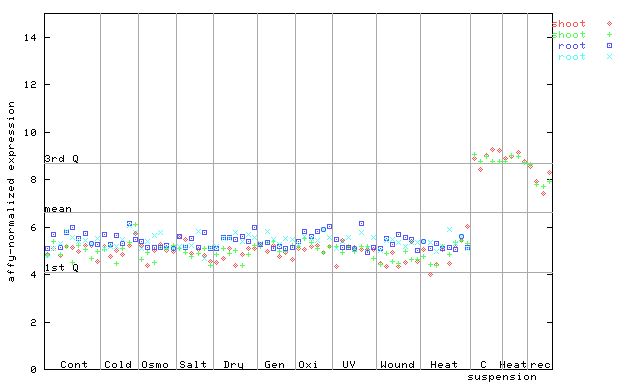
X axis is samples (pdf file), and Y axis is log2-expression. |
||||||||||||||||||||||||||||||
| AtGenExpress* (Hormone) |
257013_at
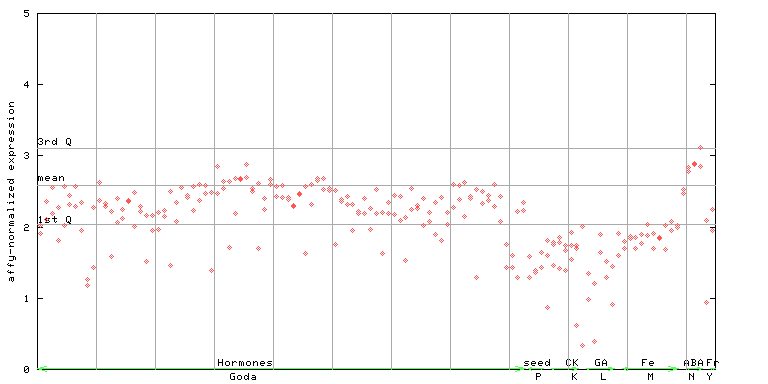
X axis is samples (xls file), and Y axis is log-expression. |
||||||||||||||||||||||||||||||
| Link to other DBs | ||
| Entrez Gene ID | 7922367 |


|
| Refseq ID (protein) | NP_001154649.1 |  |
| NP_001326309.1 |  |
|
| NP_001326310.1 |  |
|
| NP_001326311.1 |  |
|
| NP_001326312.1 |  |
|
The preparation time of this page was 0.1 [sec].
