[←][→] ath
| functional annotation | ||||||||||||||||||||||||||||||||||||||
| Function | EF-TU receptor |


|
||||||||||||||||||||||||||||||||||||
| GO BP |
|
|||||||||||||||||||||||||||||||||||||
| GO CC |
|
|||||||||||||||||||||||||||||||||||||
| GO MF |
|
|||||||||||||||||||||||||||||||||||||
| KEGG | ath04626 [list] [network] Plant-pathogen interaction (170 genes) |  |
||||||||||||||||||||||||||||||||||||
| Protein | NP_001318610.1 NP_197548.1 | |||||||||||||||||||||||||||||||||||||
| BLAST | NP_001318610.1 NP_197548.1 | |||||||||||||||||||||||||||||||||||||
| Orthologous | [Ortholog page] AT3G47090 (ath) AT3G47110 (ath) AT3G47570 (ath) AT3G47580 (ath) AT5G39390 (ath) LOC4324494 (osa) LOC4324497 (osa) LOC4324499 (osa) LOC4324501 (osa) LOC4328700 (osa) LOC4328704 (osa) LOC4328722 (osa) LOC4328724 (osa) LOC4328725 (osa) LOC4329980 (osa) LOC4329982 (osa) LOC4329983 (osa) LOC4330078 (osa) LOC4341380 (osa) LOC4342285 (osa) LOC4345065 (osa) LOC4348346 (osa) LOC4349906 (osa) LOC4350541 (osa) LOC4350749 (osa) LOC4350958 (osa) LOC7463459 (ppo) LOC7463460 (ppo) LOC7468441 (ppo) LOC7469474 (ppo) LOC7474826 (ppo) LOC7476326 (ppo) LOC7482217 (ppo) LOC7486319 (ppo) LOC7494927 (ppo) LOC9266358 (osa) LOC9267517 (osa) LOC9269230 (osa) LOC9270033 (osa) LOC9271075 (osa) LOC9271462 (osa) LOC9271745 (osa) LOC9272400 (osa) LOC11406338 (mtr) LOC11406710 (mtr) LOC11406756 (mtr) LOC11407069 (mtr) LOC11407070 (mtr) LOC11407873 (mtr) LOC11408622 (mtr) LOC11408648 (mtr) LOC11409452 (mtr) LOC11409458 (mtr) LOC11410960 (mtr) LOC11411980 (mtr) LOC11413493 (mtr) LOC11415807 (mtr) LOC11415808 (mtr) LOC11416344 (mtr) LOC11416554 (mtr) LOC11416555 (mtr) LOC11418993 (mtr) LOC11419156 (mtr) LOC11421706 (mtr) LOC11423999 (mtr) LOC11426285 (mtr) LOC11426548 (mtr) LOC11428017 (mtr) LOC11430041 (mtr) LOC11432152 (mtr) LOC11438431 (mtr) LOC25482466 (mtr) LOC25482467 (mtr) LOC25484776 (mtr) LOC25484777 (mtr) LOC25484778 (mtr) LOC25486456 (mtr) LOC25487142 (mtr) LOC25487144 (mtr) LOC25487145 (mtr) LOC25496117 (mtr) LOC25501419 (mtr) LOC100241667 (vvi) LOC100258860 (vvi) LOC100259050 (vvi) LOC100259715 (vvi) LOC100263845 (vvi) LOC100265904 (vvi) LOC100272957 (zma) LOC100776429 (gma) LOC100777490 (gma) LOC100778093 (gma) LOC100782685 (gma) LOC100783512 (gma) LOC100785561 (gma) LOC100786600 (gma) LOC100792724 (gma) LOC100801459 (gma) LOC100813523 (gma) LOC101246542 (sly) LOC101250857 (sly) LOC101251570 (sly) LOC101256934 (sly) LOC101257348 (sly) LOC101258336 (sly) LOC101261427 (sly) LOC101261723 (sly) LOC101266316 (sly) LOC102664380 (gma) LOC102664710 (gma) LOC103626075 (zma) LOC103628995 (zma) LOC103634832 (zma) LOC103634833 (zma) LOC103640781 (zma) LOC103641369 (zma) LOC103649762 (zma) LOC103650269 (zma) LOC103652739 (zma) LOC103652958 (zma) LOC103653760 (zma) LOC103653761 (zma) LOC103653766 (zma) LOC103653770 (zma) LOC103653773 (zma) LOC103654972 (zma) LOC103832809 (bra) LOC103838692 (bra) LOC103839158 (bra) LOC103841098 (bra) LOC103841563 (bra) LOC103844376 (bra) LOC103845674 (bra) LOC103848469 (bra) LOC103848471 (bra) LOC103856446 (bra) LOC103856633 (bra) LOC103857204 (bra) LOC103864894 (bra) LOC103864895 (bra) LOC103867613 (bra) LOC103868348 (bra) LOC103869459 (bra) LOC103873225 (bra) LOC103873260 (bra) LOC103873370 (bra) LOC106794146 (gma) LOC107275420 (osa) LOC107275524 (osa) LOC107276008 (osa) LOC107276079 (osa) LOC107276445 (osa) LOC107276510 (osa) LOC107276755 (osa) LOC107277108 (osa) LOC109122564 (vvi) LOC109940253 (zma) LOC109946042 (zma) LOC112422017 (mtr) LOC112422018 (mtr) LOC112422019 (mtr) LOC112422020 (mtr) LOC112937978 (osa) LOC112940090 (sly) | |||||||||||||||||||||||||||||||||||||
| Subcellular localization wolf |
|
|||||||||||||||||||||||||||||||||||||
| Subcellular localization TargetP |
|
|||||||||||||||||||||||||||||||||||||
| Gene coexpression | ||||||||||||||||||||||||||||||||||||||
| Network*for coexpressed genes |
|
|||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Coexpressed gene list |
[Coexpressed gene list for EFR] | |||||||||||||||||||||||||||||||||||||
| Gene expression | ||||||||||||||||||||||||||||||||||||||
| All samples | [Expression pattern for all samples] | |||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Development) |
246082_at
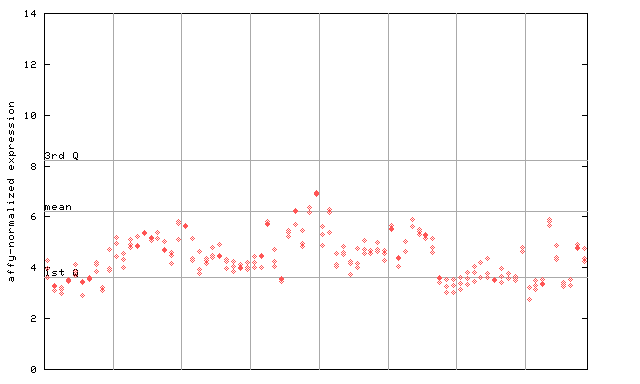
X axis is samples (pdf file), and Y axis is log2-expression. |
|||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Stress) |
246082_at
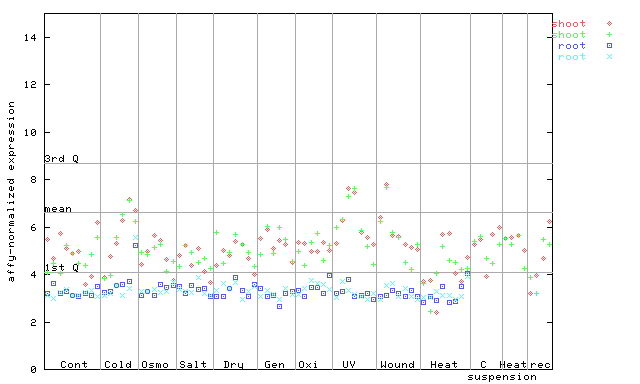
X axis is samples (pdf file), and Y axis is log2-expression. |
|||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Hormone) |
246082_at
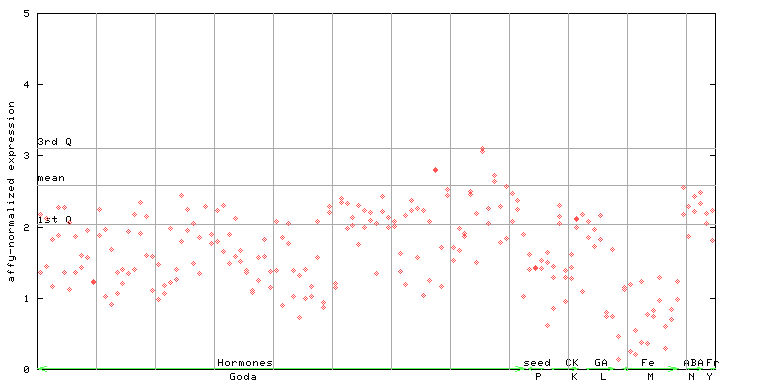
X axis is samples (xls file), and Y axis is log-expression. |
|||||||||||||||||||||||||||||||||||||
| Link to other DBs | ||
| Entrez Gene ID | 832170 |


|
| Refseq ID (protein) | NP_001318610.1 |  |
| NP_197548.1 |  |
|
The preparation time of this page was 0.3 [sec].



