[←][→] ath
| functional annotation | |||||||||||||||||||||||||||||||||||||||
| Function | SAUR-like auxin-responsive protein family |




|
|||||||||||||||||||||||||||||||||||||
| GO BP |
|
||||||||||||||||||||||||||||||||||||||
| GO CC |
|
||||||||||||||||||||||||||||||||||||||
| GO MF |
|
||||||||||||||||||||||||||||||||||||||
| KEGG | |||||||||||||||||||||||||||||||||||||||
| Protein | NP_174243.1 | ||||||||||||||||||||||||||||||||||||||
| BLAST | NP_174243.1 | ||||||||||||||||||||||||||||||||||||||
| Orthologous | [Ortholog page] SAUR75 (ath) AT1G29420 (ath) AT1G29430 (ath) SAUR63 (ath) AT1G29450 (ath) AT1G29460 (ath) AT1G29490 (ath) SAUR68 (ath) LOC4346256 (osa) LOC4347756 (osa) LOC4347757 (osa) LOC4347758 (osa) LOC4347760 (osa) LOC4347762 (osa) LOC4347763 (osa) LOC4347764 (osa) LOC4347765 (osa) LOC4347766 (osa) LOC7466808 (ppo) LOC7474969 (ppo) LOC7474970 (ppo) LOC7484612 (ppo) LOC7484613 (ppo) LOC9267694 (osa) LOC9271092 (osa) LOC18102171 (ppo) LOC18107029 (ppo) LOC25492865 (mtr) LOC100802572 (gma) LOC100803097 (gma) LOC101244143 (sly) LOC101244732 (sly) LOC101245313 (sly) LOC101252479 (sly) LOC101252771 (sly) LOC101253080 (sly) LOC101253683 (sly) LOC101253994 (sly) LOC101254589 (sly) LOC101254894 (sly) LOC101259491 (sly) LOC101259790 (sly) LOC101264565 (sly) LOC101267078 (sly) LOC101267371 (sly) LOC101267955 (sly) LOC101268244 (sly) LOC101268825 (sly) LOC103835269 (bra) LOC103835270 (bra) LOC103835271 (bra) LOC103835272 (bra) LOC103840477 (bra) LOC103840479 (bra) LOC103840483 (bra) LOC107275988 (osa) LOC107277246 (osa) LOC107278561 (osa) LOC112328596 (ppo) LOC112936358 (osa) LOC112936359 (osa) LOC123104678 (tae) LOC123104679 (tae) LOC123104686 (tae) LOC123104689 (tae) LOC123104690 (tae) LOC123104697 (tae) LOC123104698 (tae) LOC123104699 (tae) LOC123104700 (tae) LOC123104701 (tae) LOC123104702 (tae) LOC123104703 (tae) LOC123104704 (tae) LOC123104705 (tae) LOC123106336 (tae) LOC123107613 (tae) LOC123107616 (tae) LOC123107622 (tae) LOC123107623 (tae) LOC123107624 (tae) LOC123107625 (tae) LOC123107626 (tae) LOC123107627 (tae) LOC123107628 (tae) LOC123112943 (tae) LOC123112946 (tae) LOC123112947 (tae) LOC123112951 (tae) LOC123112953 (tae) LOC123112954 (tae) LOC123112963 (tae) LOC123112968 (tae) LOC123112972 (tae) LOC123112973 (tae) LOC123112974 (tae) LOC123112977 (tae) LOC123112978 (tae) LOC123112980 (tae) LOC123112981 (tae) LOC123112982 (tae) LOC123116771 (tae) LOC123116772 (tae) LOC123116774 (tae) LOC123116777 (tae) LOC123116778 (tae) LOC123116779 (tae) LOC123116780 (tae) LOC123116781 (tae) LOC123120838 (tae) LOC123120845 (tae) LOC123120846 (tae) LOC123122456 (tae) LOC123122457 (tae) LOC123122464 (tae) LOC123122465 (tae) LOC123122467 (tae) LOC123122468 (tae) LOC123122469 (tae) LOC123122470 (tae) LOC123122474 (tae) LOC123122478 (tae) LOC123122479 (tae) LOC123122485 (tae) LOC123122487 (tae) LOC123122488 (tae) LOC123122490 (tae) LOC123122492 (tae) LOC123122493 (tae) LOC123122494 (tae) LOC123122495 (tae) LOC123122496 (tae) LOC123122498 (tae) LOC123122499 (tae) LOC123122500 (tae) LOC123125334 (tae) LOC123125347 (tae) LOC123152558 (tae) LOC123152559 (tae) LOC123160149 (tae) LOC123163909 (tae) LOC123398943 (hvu) LOC123411073 (hvu) LOC123451780 (hvu) LOC123451783 (hvu) LOC123451789 (hvu) LOC123451799 (hvu) LOC123451800 (hvu) LOC123451810 (hvu) LOC123451811 (hvu) LOC123451815 (hvu) LOC123451816 (hvu) LOC123451817 (hvu) LOC123451818 (hvu) LOC123451819 (hvu) LOC123451820 (hvu) LOC123451821 (hvu) LOC123451822 (hvu) LOC123451823 (hvu) LOC123451824 (hvu) LOC123451826 (hvu) LOC123451827 (hvu) LOC123451828 (hvu) LOC123451830 (hvu) LOC123451831 (hvu) LOC123451832 (hvu) | ||||||||||||||||||||||||||||||||||||||
| Subcellular localization wolf |
|
||||||||||||||||||||||||||||||||||||||
| Subcellular localization TargetP |
|
||||||||||||||||||||||||||||||||||||||
| Gene coexpression | |||||||||||||||||||||||||||||||||||||||
| Network*for coexpressed genes |
|
||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Coexpressed gene list |
[Coexpressed gene list for AT1G29500] | ||||||||||||||||||||||||||||||||||||||
| Gene expression | |||||||||||||||||||||||||||||||||||||||
| All samples | [Expression pattern for all samples] | ||||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Development) |
259773_at
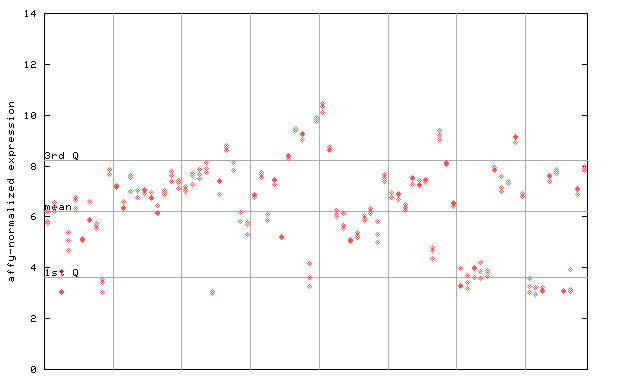
X axis is samples (pdf file), and Y axis is log2-expression. |
||||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Stress) |
259773_at
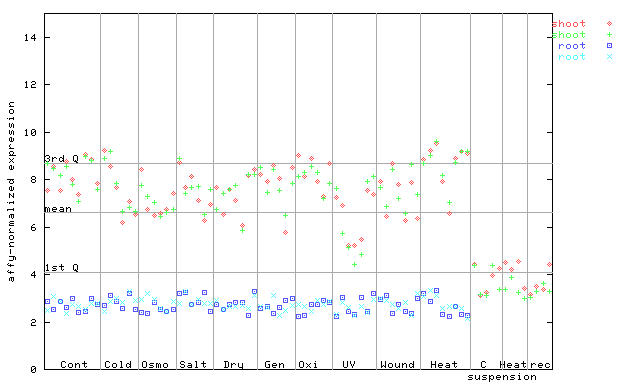
X axis is samples (pdf file), and Y axis is log2-expression. |
||||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Hormone) |
259773_at
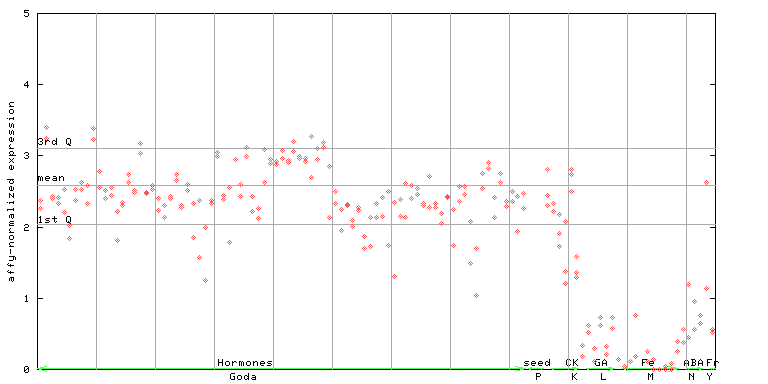
X axis is samples (xls file), and Y axis is log-expression. |
||||||||||||||||||||||||||||||||||||||
| Link to other DBs | ||
| Entrez Gene ID | 839827 |


|
| Refseq ID (protein) | NP_174243.1 |  |
The preparation time of this page was 0.4 [sec].


