[←][→] ath
| functional annotation | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Function | cytochrome P450, family 72, subfamily A, polypeptide 13 |




|
||||||||||||||||||||||||||||||||||||||||||||||||||||
| GO BP |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| GO CC |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| GO MF |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| KEGG | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Protein | NP_001326383.1 NP_001326384.1 NP_188084.1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| BLAST | NP_001326383.1 NP_001326384.1 NP_188084.1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Orthologous | [Ortholog page] CYP72A7 (ath) CYP72A8 (ath) CYP72A9 (ath) CYP72A10 (ath) CYP72A11 (ath) CYP72A14 (ath) CYP72A15 (ath) LOC4323921 (osa) LOC4324603 (osa) LOC4324604 (osa) LOC4324605 (osa) LOC4326657 (osa) LOC4326659 (osa) LOC4326660 (osa) LOC4326661 (osa) LOC4326663 (osa) LOC4326694 (osa) LOC4338594 (osa) LOC7462323 (ppo) LOC7462324 (ppo) LOC9272564 (osa) LOC11434248 (mtr) LOC11435500 (mtr) LOC18103298 (ppo) LOC18103304 (ppo) LOC18103309 (ppo) LOC18103313 (ppo) LOC18103316 (ppo) LOC18103318 (ppo) LOC18109634 (ppo) LOC25486186 (mtr) LOC25486774 (mtr) LOC25486777 (mtr) LOC25487123 (mtr) LOC25487124 (mtr) LOC25487125 (mtr) LOC25487128 (mtr) LOC25487130 (mtr) LOC25487132 (mtr) LOC100777623 (gma) LOC100780475 (gma) LOC100780780 (gma) LOC100781923 (gma) LOC100782454 (gma) LOC100782998 (gma) CYP72A67 (gma) CYP72A69 (gma) LOC100808323 (gma) LOC100808851 (gma) LOC100809929 (gma) LOC101245896 (sly) LOC101248145 (sly) LOC101248428 (sly) LOC101252201 (sly) LOC101255312 (sly) LOC101255604 (sly) LOC101256104 (sly) LOC101262533 (sly) LOC101263131 (sly) LOC101263430 (sly) LOC101263726 (sly) LOC101264341 (sly) LOC101264644 (sly) LOC101264947 (sly) LOC101265252 (sly) LOC101265559 (sly) LOC101265840 (sly) CYP72A14 (sly) GAME7 (sly) LOC101268591 (sly) LOC102667924 (gma) LOC103841861 (bra) LOC103841876 (bra) LOC103841893 (bra) LOC103859585 (bra) LOC103859587 (bra) LOC103859588 (bra) LOC103870002 (bra) LOC103870003 (bra) LOC103870005 (bra) LOC103870006 (bra) LOC103870007 (bra) LOC103870009 (bra) LOC103871394 (bra) LOC108871396 (bra) LOC123041114 (tae) LOC123049127 (tae) LOC123050315 (tae) LOC123058385 (tae) LOC123061542 (tae) LOC123061545 (tae) LOC123063983 (tae) LOC123065452 (tae) LOC123065453 (tae) LOC123065454 (tae) LOC123067043 (tae) LOC123067045 (tae) LOC123070179 (tae) LOC123074553 (tae) LOC123078619 (tae) LOC123078621 (tae) LOC123081026 (tae) LOC123081845 (tae) LOC123082514 (tae) LOC123083092 (tae) LOC123094942 (tae) LOC123099300 (tae) LOC123100014 (tae) LOC123102416 (tae) LOC123125828 (tae) LOC123130912 (tae) LOC123131290 (tae) LOC123134472 (tae) LOC123134954 (tae) LOC123139478 (tae) LOC123141380 (tae) LOC123141751 (tae) LOC123146064 (tae) LOC123148942 (tae) LOC123148943 (tae) LOC123154211 (tae) LOC123156436 (tae) LOC123156437 (tae) LOC123158869 (tae) LOC123166482 (tae) LOC123167937 (tae) LOC123169547 (tae) LOC123169575 (tae) LOC123179755 (tae) LOC123179756 (tae) LOC123182723 (tae) LOC123186319 (tae) LOC123402192 (hvu) LOC123403395 (hvu) LOC123411806 (hvu) LOC123417826 (hvu) LOC123428321 (hvu) LOC123431001 (hvu) LOC123439993 (hvu) LOC123439994 (hvu) LOC123441197 (hvu) LOC123443923 (hvu) LOC123446623 (hvu) | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Subcellular localization wolf |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| Subcellular localization TargetP |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| Gene coexpression | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Network*for coexpressed genes |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Coexpressed gene list |
[Coexpressed gene list for CYP72A13] | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| Gene expression | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| All samples | [Expression pattern for all samples] | |||||||||||||||||||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Development) |
258114_at
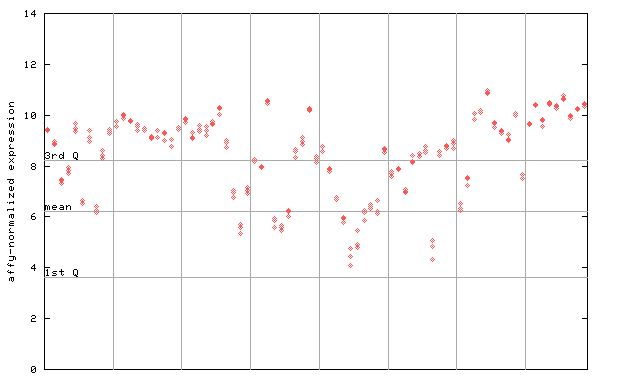
X axis is samples (pdf file), and Y axis is log2-expression. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Stress) |
258114_at
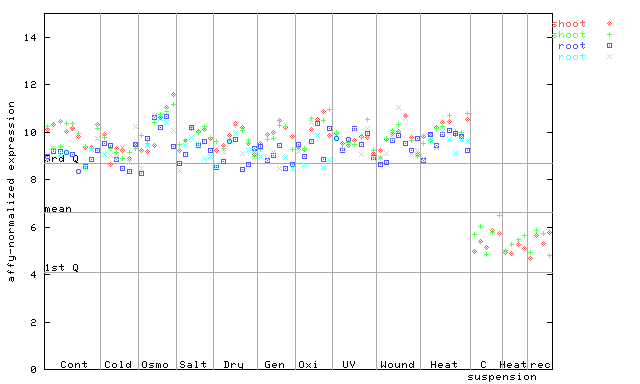
X axis is samples (pdf file), and Y axis is log2-expression. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| AtGenExpress* (Hormone) |
258114_at
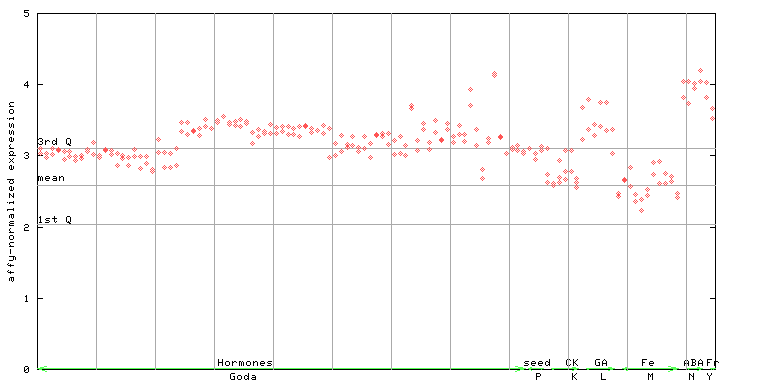
X axis is samples (xls file), and Y axis is log-expression. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| Link to other DBs | ||
| Entrez Gene ID | 820694 |


|
| Refseq ID (protein) | NP_001326383.1 |  |
| NP_001326384.1 |  |
|
| NP_188084.1 |  |
|
The preparation time of this page was 0.1 [sec].


